Entendiendo los métodos Markov Chain Monte Carlo
Una de las cosas más atractivas de la inferencia bayesiana es que permite la incorporación de conocimiento previo o creencias mediante distribuciones de probabilidad sobre los parámetros de interés. Por ejemplo, uno querría estimar cuál es la tasa de letalidad de una enfermedad y querría incorporar la información de un estudio previo, que indica que es del 0.5%. Esto confronta con la escuela frecuentista de la estadística, que asume que las probabilidades están estrictamente relacionadas con la frecuencia de los eventos. Para los frecuentistas, el valor real de este parámetro (por ej, la tasa de letalidad) es fijo y desconocido y, por tanto, no tiene sentido establecer ninguna distribución de probabilidad sobre él.
Esto se traduce en que, por ejemplo, los intervalos de confianza de la estadística frecuentista tengan una interpretación que a muchos no les parece tan natural. Por ejemplo, para el caso de la letalidad, diríamos que nuestro intervalo al 90% de confianza contendrá en un 90% de las veces al valor real de la tasa de letalidad. En otras palabras, si tomamos 100 muestras independientes y calculamos el intervalo de confianza para cada una de ellas, la esperanza es que 90 de cada 100 contendrían el valor real. Esta es una excelente visualización que muestra lo anterior.
Para muchos, sin embargo, es más natural preguntarse: ¿cuál es la probabilidad de que mi parámetro esté dentro de un cierto intervalo? Esto es posible bajo el prima bayesiano. En la concepción bayesiana de la estadística se acepta que, dado que nuestro conocimiento sobre el valor real del parámetro en cuestión es limitado, tiene sentido establecer distribuciones de probabilidad sobre él. La tasa de letalidad puede ser del 0.4% con una probabilidad \(P_1\), 0.5% con una probabilidd \(P_2\), etc.
Inferencia bayesiana: problemas y soluciones
La estadística bayesiana, se basa en el Teorema de Bayes:
En donde \(P(\theta | x)\) es la probabilidad a posteriori del parámetro dados los datos \(x\) (esto es lo que queremos estimar), \(P(x|\theta)\) es la función de verosimilitud e indica la probabilidad de observar \(x\) dado \(\theta\) y \(P(\theta)\) refleja la creencia a priori sobre nuestro parámetro \(\theta\). El denominador, que en la versión discreta sería \(P(x) = \sum_{\theta}P(x,\theta)\) (se le suele denominar la evidencia), es precisamente el elemento que complica el cálculo de la ecuación. La integral que se ve en la fórmula puede complicarse de tal forma que hace muy difícil su cálculo de manera analítica. Aquí es donde entran en juego los métodos numéricos como Markov Chain Monte Carlo (MCMC). Dentro de los MCMC, hay diversos métodos, pero aquí el que voy a tratar es el algoritmo Metropolis-Hastings.
Veamos un ejemplo concreto:
En primer lugar, simulemos algunos datos. En concreto, 50 valores de una distribución de Poisson con media 5.
df <- rpois(n = 50, lambda = 5)
barplot(table(df), main = 'Data distribution', ylab = 'Count', space = 0.2)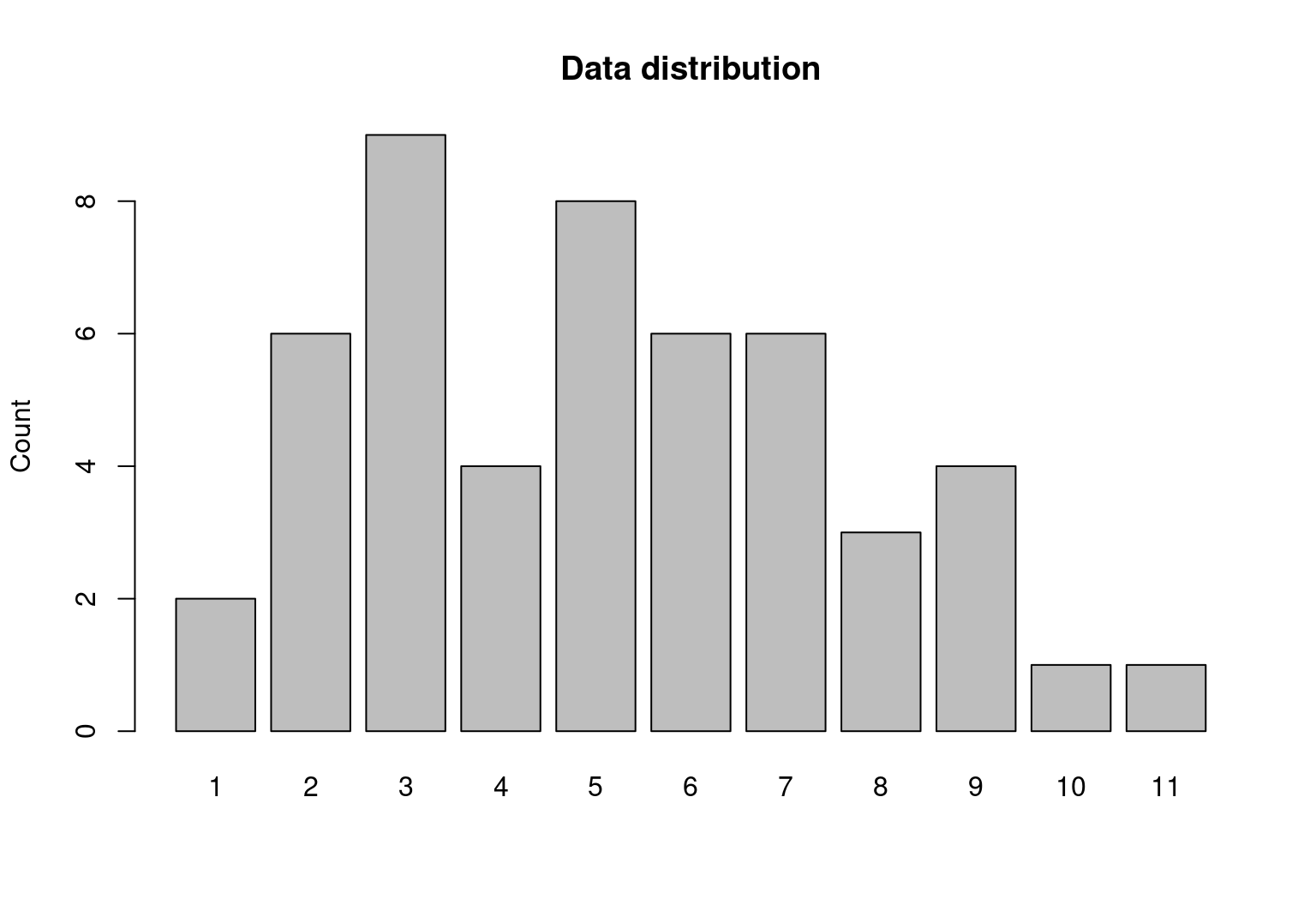
El segundo paso es definir el modelo, que en este caso, como hemos generado los datos bajo una Poisson, la verosimilitud también será una Poisson con parámetro \(\lambda\). Por último, tenemos que fijar una distribución a priori para nuestro parámetro \(\lambda\) de interés. En este caso, nuestra distribución a priori será una Gamma con parámetros de forma \(\alpha\) (shape) y \(\beta\) (rate) tal que su función de densidad:
Para analizar este modelo de forma analítica es conveniente que la distribución a priori sea una Gamma, ya que la distribución a priori Gamma para \(\lambda\) es conjugada (las distribuciones a priori y posteriori coinciden), es decir, que nuestra distribución a posteriori también será una Gamma. Los parámetros de esta distribución Gamma a posteriori son:
Esta distribución a posteriori se ha podido calcular de forma analítica porque las distribuciones a priori y posteriori son conjugadas, pero esto no siempre es el caso, de ahí que se requiera de métodos numéricos que solucionen este problema.
Algoritmo Metropolis-Hastings
El funcionamiento del algoritmo es el siguiente:
- Comenzamos con un valor inicial aleatorio de \(\lambda\), en este caso, 3.
- Saltamos a otro punto cualquiera. En este caso, bajo una distribución normal con el valor actual de lambda \((\lambda_a)\) como media y distribución típica fijada a 1. Este será nuestro nuevo lambda propuesto, \(\lambda_p\)
- Calculamos la verosimilitud para el valor actual y propuesto de \(\lambda\), tal que \(L(\lambda|X_1..X_n)=\prod_{i=1}^{N}\frac{e^{-\lambda}\lambda^{x_i}}{X_i!}\)
- Calculamos las probabilidades a posteriori como el producto entre la verosimilitud y la distribución a priori. Después, calculamos el ratio de aceptación, que se obtiene al dividir las probabilidades a posteriori del lambda propupuesto \((\lambda_p)\) y el lambda actual \((\lambda_a)\), consiguiendo así cancelar la evidencia, \(P(x)\), que es el término del que precisamente queríamos deshacernos:
- Si este ratio es mayor a un valor aleatorio entre 0 y 1, aceptamos la propuesta y tomamos \(\lambda_a = \lambda_p\). En caso contrario, mantenemos el valor actual, \(\lambda_a\). La lógica es la siguiente, a través del ratio de aceptación lo que estamos consiguiendo es que estamos recorriendo con mayor frecuencia las regiones con mayor probabilidad a posteriori en detrimento de oras con menor probabilidad.
- Repetimos \(N\) veces.
En código, sería así:
mcmc <-
function(iters, lambda_ini = 3, shape_prior = 5, rate_prior = 2) {
# Valores iniciales
lambda_a <- lambda_ini
posterior <- lambda_a
# Iteramos n veces
for (i in seq_len(iters)) {
# Elegimos un nuevo candidato a partir de una distirbución normal
# con actual valor de lambda como media y una sd de 1
lambda_p <- rnorm(n = 1, mean = lambda_a, sd = 1)
# Calculamos la verosimilitud
likelihood_a <- prod(dpois(x = df, lambda_a))
likelihood_p <- prod(dpois(x = df, lambda_p))
# Calculamos la probabilidad a priori de los valores actuales y propuestos
prior_a <- dgamma(x = lambda_a, shape = shape_prior, rate = rate_prior)
prior_p <- dgamma(x = lambda_p, shape = shape_prior, rate = rate_prior)
# Probabilidad a posteriori
p_current <- likelihood_a * prior_a
p_proposal <- likelihood_p * prior_p
# Ratio de aceptacion
p_accept <- p_proposal / p_current
# Si el rario es mayor a un valor arbitrario entre 0 y 1, aceptamos propuesta
accept <- runif(1, 0, 1) < p_accept
if (accept) lambda_a <- lambda_p
posterior <- c(posterior, lambda_a)
}
posterior
}Veamos los resultados:
set.seed(1993)
# Valores a priori de gamma (tambien definidos en la funcion)
shape_prior = 5
rate_prior = 2
# Corremos el algoritmo MH
tst <- mcmc(iters = 10000)
# funcion de densidad
dd <- density(tst)
plot(dd, main = 'Analytical and MCMC-based posterior', col = "steelblue", lwd = 1)
# Distribucion gamma analitica a posterori
lines(x = dd$x, y = dgamma(dd$x,
shape = sum(df) + shape_prior,
rate = rate_prior + length(df)),
type = 'l',
col = "darkred",
lty = 2,
lwd = 1.5)
legend(3, 1.3,
legend = c("Simulated MCMC","Analytical posterior"),
col = c("steelblue","darkred"),
lty = c(1, 2),
lwd = c(1.5, 1.5),
box.lty = 0)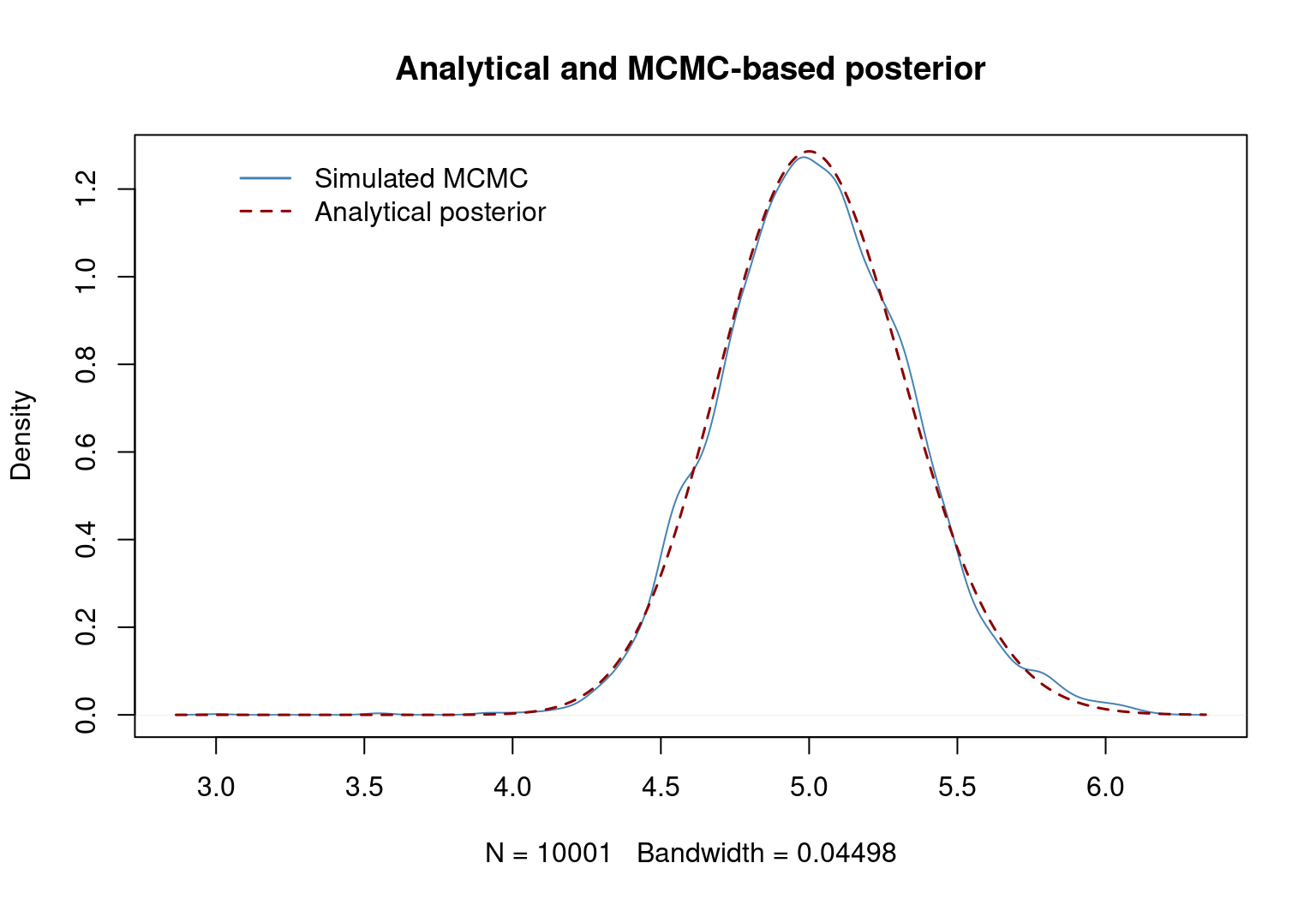
Finalmente, veamos la convergencia del algoritmo al valor de \(\lambda\) esperado.
mcmc_acum <- vector(mode = 'numeric')
for (i in seq(from = 0, to = 500, by = 1)) {
mcmc_acum <- c(mcmc_acum, mean(mcmc(i, lambda_ini = 12)))
}
plot(x = seq_along(mcmc_acum),
y = mcmc_acum,
ylab = 'lambda',
type = 'l',
xlab = 'Iterations',
main = 'Convergence diagnosis')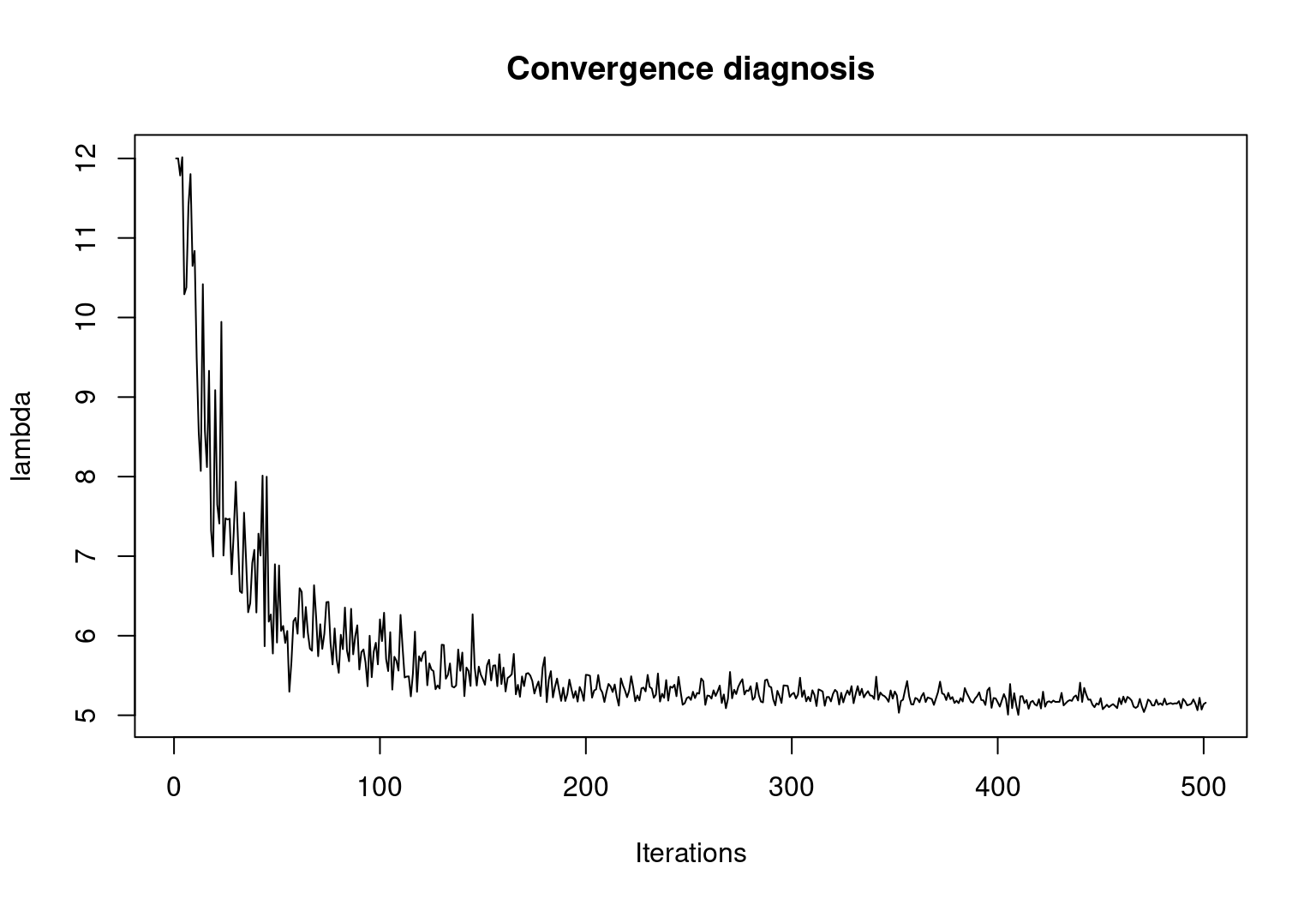
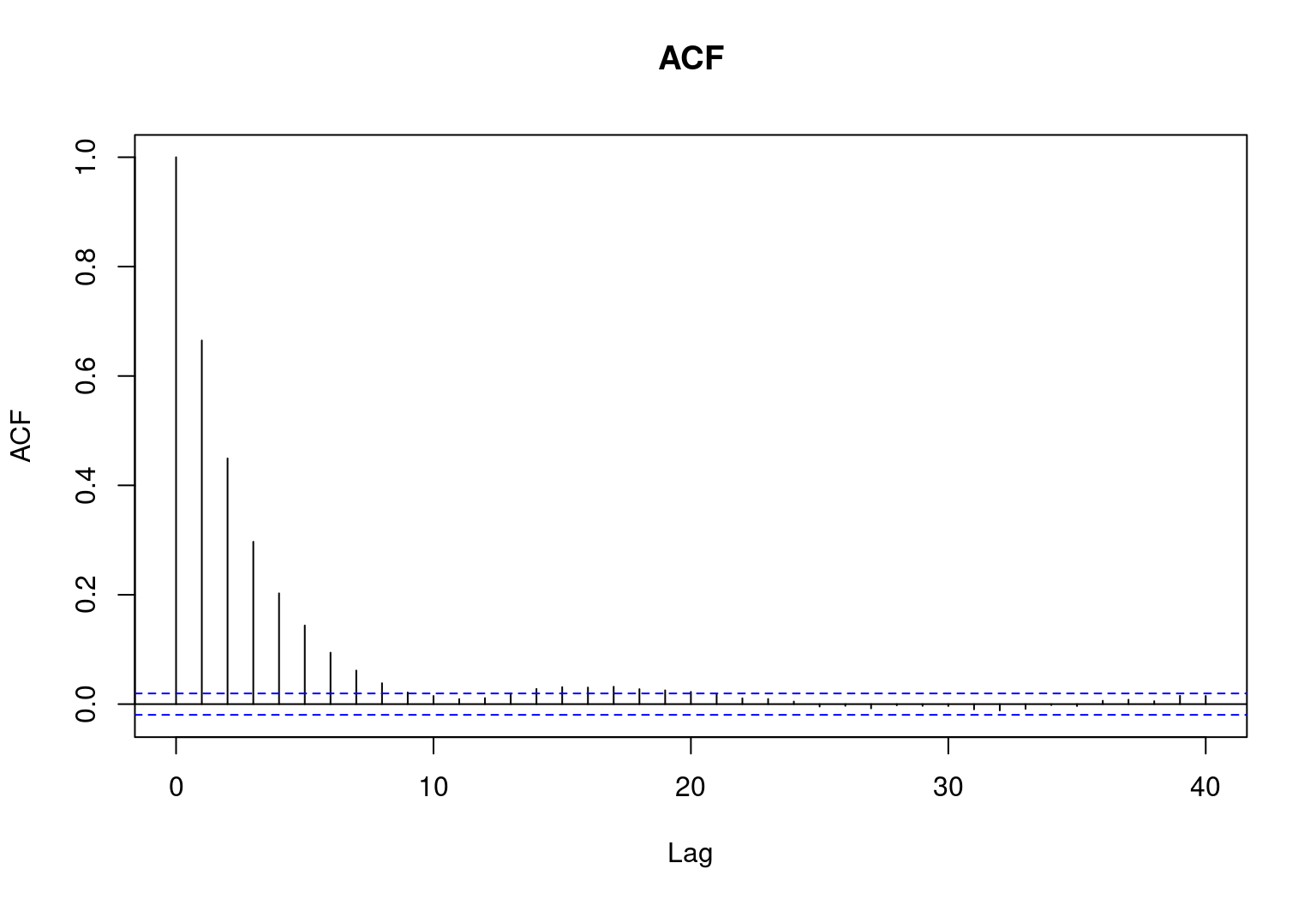
acf(tst, main = 'ACF')